Nicht erst seit Angelina Jolies New York Times-Artikel über die Konsequenzen die sie aus ihrem Gentest gezogen hat gibt es Interesse an Gen-Tests in der Quantified Self-Community und 23andMe. Während in medizinischem Rahmen durchgeführte Tests nicht ohne weiteres zu bekommen sind, gibt es seit circa 2006 auch sogenannte Direct-To-Consumer (DTC) Gentests, welche direkt an Endkunden vertrieben. Wie funktionieren solche Tests? Und was kann der geneigte Self Quantifier daraus lernen?
Anders als medizinisch-diagnostische Tests, wie die von Jolie beschriebenen Tests für Mutationen in den BRCA-Genen, untersuchen DTC Gentests eine größere Anzahl von genetischen Markern die über das gesamte Genom verteilt sind. Ohne dabei jedoch das gesamte Genom zu sequenzieren.

Größter Anbieter für solche DTC-Tests – die als Hilfe zur Weiterbildung und nicht für die medizinische Diagnostik gedacht sind – ist 23andMe mit Sitz im Silicon Valley. Für ca. $100 zuzüglich des weltweiten Versands (~ $70 nach Deutschland) bekommt man dort knapp 1 Million Marker seines Genoms ausgewertet.
Der eigentliche Bestellvorgang ist dabei nicht groß anders als eine Bestellung bei Amazon. Nach ein paar Klicks auf der Webseite und der Bezahlung mit Kreditkarte macht sich ein Paket per Express-Versand von Kalifornien aus auf den Weg nach Deutschland. In dem Paket befindet sich neben einem schon adressierten Paket für den Rückversand ein kleines Probenröhrchen in dem der eigene Speichel gesammelt wird. Nachdem das Röhrchen gefüllt ist reicht ein Anruf bei DHL und die biologische Probe macht sich per Express zurück auf den Weg nach Kalifornien. Bei mir hat es von Bestelleingang bis zur Bestätigung das mein Speichel in Kalifornien eingegangen ist ~5 Tage gedauert. Danach beginnt das eigentlich warten, dann es kann 4-6 Wochen dauern bis die Testergebnisse über die 23andMe-Webseite zur Verfügung stehen.
Die Marker die 23andMe testet, die sogenannten Single Nucleotide Polymorphisms – oder kurz SNPs – bezeichnen Positionen im menschlichen Genom von denen bekannt ist das sie, anders als der Großteil des gesamten Genoms, variabel ausgeprägt sein können zwischen zufällig betrachteten Menschen. Für viele dieser Marker ist bekannt ob, beziehungsweise das, sie mit der Ausprägung von bestimmten Eigenschaften in Verbindung stehen. Die möglichen Eigenschaften reichen dabei von Dingen wie der Haar- und Augenfarbe bis zu Dingen wie dem Risiko an Alzheimer zu erkranken.
Gefunden werden solche Assoziationen durch den Einsatz von sogenannten Genomweiten Assoziationsstudien (kurz GWAS). Bei diesen Studien vergleicht man zwei verschiedene Gruppen: Eine Gruppe zeigt die Eigenschaft an der man interessiert ist, während der Kontrollgruppe eben diese Eigenschaft fehlt. Für beide Gruppen macht man dann solche SNP-basierten Gentests und vergleicht anschliessend die Ergebnisse zwischen beiden Gruppen. Findet man jetzt eine bestimmte Ausprägung eines SNPs in einer der beiden Gruppen überrepräsentiert, so kann man einen Link zwischen der Eigenschaft und dem SNP herstellen.
Ein konkretes Beispiel: Bei Assoziationsstudien wurde der Marker rs1447295 in der Ausprägung „A“ deutlich häufiger in der Population der Prostatakrebs-Patienten gefunden als in der gesunden Kontroll-Population, in der vor allem die Marker-Ausprägung „C“ gefunden wurde (Die Namen der möglichen Ausprägungen, auch Allele genannt, stammen aus dem 4-Buchstaben-Alphabet in dem unser, und jedes andere, Genom geschrieben ist).
Diese Fundhäufigkeiten kann man dann umrechnen darauf wie groß die Wahrscheinlichkeit ist, das man tatsächlich in die entsprechende Population fällt, wenn man selbst eine überrepräsentierte Variante trägt. Für das Prostatakrebs-Beispiel: Wer zwei mal das Allel „A“ an Marker rs1447295 trägt, der hat eine doppelt so hohe Wahrscheinlichkeit in die Prostatakrebs-Gruppe zu gehören wie der Durchschnitt.
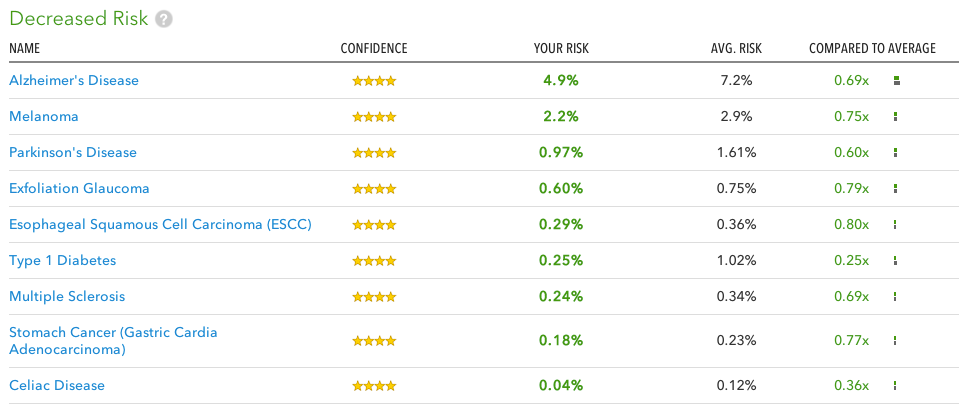
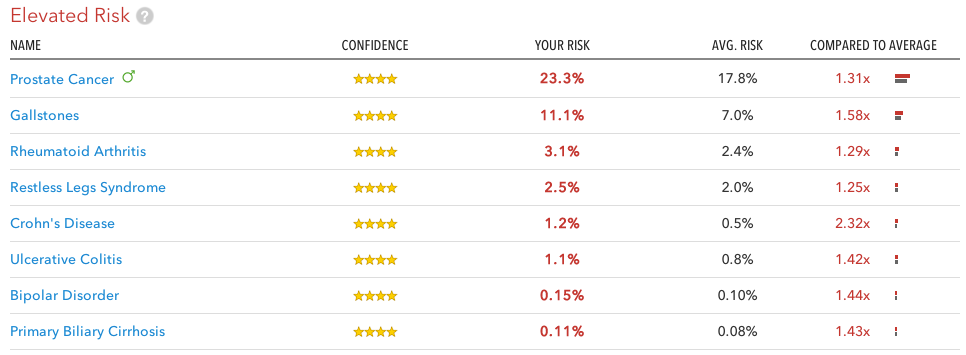
So einfach diese Studien im Grundprinzip auch sind, hier zeigt sich schon die Limitierung: Wirkliche Schwarz/Weiss-Ergebnisse darf man sich von einem Gentest der auf GWAS setzt nicht erhoffen, stattdessen betritt man ein Land voller erhöhter und erniedrigter Wahrscheinlichkeiten. So hat 23andMe für über 200 verschiedene Krankheiten und andere Eigenschaften die vorhandene wissenschaftliche Literatur gewälzt und liefert darauf basierende Auswertungen an seine Kunden zurück.
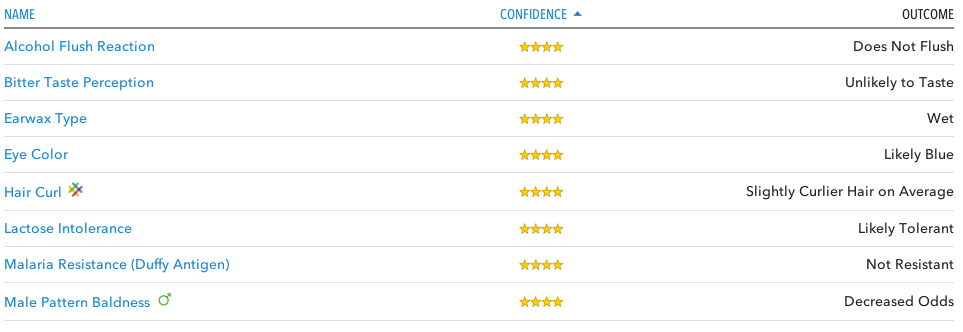
Sortiert werden die Auswertungen in verschiedene Kategorien: Krankheitsbezogene Vorhersagen darüber ob man ein erhöhtes, typisches oder erniedrigtes Risiko für sie hat; wie gut und effektiv man Medikamente verträgt; und eine allgemeine Kategorie in die Dinge wie die Konsistenz des Ohrenschmalz, Laktose(in)toleranz oder Haarausfall. Dazu gibt es noch eine ganze Reihe an Informationen zur Abstammung, wer also auf Suche nach Ahnen gehen will, oder schon immer wissen wollte zu wie viel Prozent er Neandertaler ist kann auch das tun.
Für die Risiko-Vorhersagen bezüglich Krankheiten und Medikamentenverträglichkeit gibt 23andMe auch mit an für wie Vertrauenswürdig sie die bisherigen Studien zu diesen Assoziationen halten. Mit einem einfachen Vier-Sterne-Rating ähnlich zu Amazon bekommt man einen schnellen Überblick darüber wie gesichert die Angaben sind. Vertrauenswürdige (4-Sterne) Assoziationen wurden in mehreren unabhängigen Studien gefunden und in Assoziationsstudien mit großer Stichprobengröße gefunden. Nur für diese 4-Sterne Fälle gibt 23andMe dann auch ein Krankheitsrisiko in Prozent an.
Wem die Auswertungen von 23andMe nicht reichen, der kann seine Rohdaten auch dort herunterladen und in andere Tools, wie die Promethease von *SNPedia* oder in *openSNP* einspeisen und dort zusätzliche Auswertungen zu seinem Genom bekommen.
Die *SNPedia* ist ein Wiki in dem jeder mitwirken kann um die wissenschaftliche Fach-Literatur – über die genetischen Variationen die von Anbietern wie 23andMe getestet werden – zu katalogisieren um so einen schnellen Überblick zu ermöglichen. Diese, auch maschinenlesbaren, Zusammenfassungen werden von Promethease genutzt um Reports ganz ähnlich zu denen von 23andMe zu produzieren. Promethease lässt sich kostenlos verwenden, allerdings dauert die Erstellung des persönlichen Reports dann einige Zeit. Wer es eilig hat (oder mehr Features will) kann auch die Premium-Version nutzen.
*openSNP* ist vor allem ein Platz an dem Benutzer ihre DTC Gentests in die Public Domain veröffentlichen können, um sie so der Allgemeinheit zur Verfügung zu machen um z.B. eigene Assoziationsstudien durchzuführen. Gleichzeitig bietet openSNP aber auch eine Übersicht über Fachliteratur über und bekannte Auswirkungen der einzelnen Markern. Dazu werden verschiedene Datenquellen automatisch angezapft: Fachliteratur wird aus der Datenbank des Anbieters Mendeley gesucht, sowieso aus den frei für alle zugänglichen Fachzeitschriften der *Public Library of Science*, zur Vorhersage der Auswirkungen von einzelnen Markern werden neben der *SNPedia* auch die Datenbanken des *Personal Genome Projects* und des *National Human Genome Research Institute* durchsucht.
Aber was kann man nun mit den Ergebnissen anfangen? Wie schon gesehen bieten DTC Gentests nach Art von 23andMe meistens keine „harten“ Fakten, sondern Wahrscheinlichkeiten. Und je nach Art der Krankheit für die ein erhöhtes Risiko besteht kann man auch wenig für die Vorsorge unternehmen. In anderen Fällen kann eine vom Gentest gegebene Prognose durchaus dazu genutzt werden seinen Lebenswandel entsprechend anzupassen und Vorsorgeuntersuchungen wahrzunehmen.
Mir persönlich hat der Gentest ein paar spannende Dinge gezeigt: Zum einen bin ich für keine der direkt vererbbaren Krankheiten die 23andMe testet Überträger. Mangels akutem Kinderwunsch ist das zwar nicht von besonderer Relevanz, aber nett zu wissen. Außerdem habe ich typische bzw. erniedrigte Risiken für Alzheimer und Parkinson’s, was – mangels Heilungsmöglichkeiten zur Zeit – beruhigend ist. Als jemand mit schnellen Koffein-Metabolismus könnte es sogar sein das regelmässiger Kaffee-Genuss mein Herzinfarktsrisiko senkt, jetzt habe ich also sogar eine gute Ausrede wenn ich morgens zur Tasse greife. Für mich das Spannendste: Mein Prostatakrebs-Risiko liegt bei knapp 25%. Und nachdem ich mir meine Rohdaten angeschaut habe kam ich zu dem Schluss das mein Vater ein vergleichbar hohes Risiko haben sollte, was ihn schlussendlich dazu überredet hat mal zur Vorsorge zu gehen. Die dann auch tatsächlich einen Tumor zu Tage gefördert hat der erfolgreich behandelt wurde.
Ob sich ein Gentest generell lohnt? Das muss jeder für sich entscheiden und es hängt auch nicht unmassgeblich von den Ergebnissen ab die man präsentiert bekommt. Wer mit seinen Rohdaten, die bei einem solchen Test auch abfallen, rumspielen will sollte sich allerdings auf eine relativ steile Lernkurve einstellen: Während Anbieter wie 23andMe die Testergebnisse auch für Laien verständlich aufbereiten bzw. es zumindest versuchen, sind viele andere Informationsquellen nicht direkt darauf eingestellt und erfordern eine nähere Beschäftigung mit der Materie. Eins sollte jeder der einen solchen Test macht aber in keinem Fall vergessen: Ein erhöhtes oder erniedrigtes Risiko ist weder ein Todesurteil noch eine Entwarnung auf Lebenszeit.
Dies ist ein Gastbeitrag von Bastian Greshake, Doktorand in der angewandten Bioinformatik der Universität Frankfurt. Erst ist Mitgründer des Portals openSNP, welches Gentest-Ergebnisse in die Public Domain überführt und zusammen mit Auswertungen verfügbar macht. Außerdem bloggt er über seine Erfahrung mit dem Quantified Self und aus der Wissenschaft.
igrowdigital Newsletter
Diese Inhalte sind gut für Dich und Deine Gesundheit